目次
- 1 CRISPR/Cas9の発明に2020年ノーベル化学賞
- 2 祝☆ノーベル化学賞2020
- 3 CRISPR技術の説明
- 4 CRISPRの発見と応用技術の進展のタイムライン
- 5 CRISPR-Casシステムの作用機序と生物学的な意味の解明
- 6 CRISPRの遺伝子編集技術への応用
- 7 CRISPRの発見者は誰?
- 8 ゲノム編集技術CRISPR/Cas9の特許をめぐる熾烈な争い
- 9 CRISPRの発見からツールとしての利用まで
- 10 CRISPR関連の略語、用語
- 11 遺伝子編集技術CRISPR-Casシステムの技術的な問題点
- 12 CRIPR-Casの種類と分類
- 13 バイオ関連企業による研究者向けのCRISPR-Cas技術解説
- 14 レビュー論文
- 15 原著論文
- 16 参考
CRISPR/Cas9の発明に2020年ノーベル化学賞
2020年10月7日のノーベル財団の発表によりますと、2020年のノーベル化学賞が、Jennifer A. Doudna(ジェニファー・ダウドナ)、 Emmanuel Charpentier(エマニュエル・シャルパンティエ)の両氏に与えられることになりました。

Announcement of the 2020 Nobel Prize in Chemistry Nobel Prize
祝☆ノーベル化学賞2020
ノーベル賞の枠は3人。CRISPR/Cas9の有用性は直ちに認識されて熾烈な特許戦争が勃発していたわけですが、Jennifer A. Doudna(ジェニファー・ダウドナ)、 Emmanuel Charpentier(エマニュエル・シャルパンティエ)の二人がノーベル賞3人枠の中に入ることは、誰の目にも明らかでした。ノーベル賞をもらって当然と言われてきましたが、それでも、実際に本当に授賞が決まるとこれほど嬉しいものなんだなあとこの映像を見ていて思いました。
First Day in a Nobel Life: Jennifer Doudna 2020/10/07 UC Berkeley
別にノーベル賞をもらうために研究をするわけではありませんが、研究が正当に認めらることは研究者にとって非常に大事な部分なのだと思います。
ゲノム編集技術の革命的なツールであるCRISPR/Cas9(くりすぱー きゃすないん)ですが、もとはといえば、自然界においてバクテリアがウイルスから身を守るために備えているシステムです。2012年にこれがゲノム編集のツールとして利用できることが示され、わずか数年の間に、この技術を使わないラボがあるのか?というくらいにまで、研究の世界の隅々にまで浸透しました。
CRISPR技術の説明
適度な詳しさで、CRISPRをわかりやすく簡潔に説明した動画。
What is CRISPR?
- 総説 ゲノムから見た最近の進化ーCRISPによる生存戦略ー 中川一路 Dental Medicine Research 33(3):236-241. (2013). PDFリンク 図1(A)CRISPRによる外来性因子の取り込みの機構。(2)CRISPRによる外来性因子の認識と排除
CRISPRの発見と応用技術の進展のタイムライン
CRISPR/Casが働く仕組みの解明には多くの研究者が関与しており、メジャーな発見だけでも論文が多数になるため、時系列にまとめたサイトを紹介しておきます。
- CRISPR TIMELINE (BROAD INSTITUTE)
- CRISPR Timeline (CRIPR Update)
CRISPRの発見
大阪大学微生物病研究所の研究グループが奇妙な繰り返し配列を大腸菌のゲノムで見つけ、石野 良純(いしの よしずみ)博士らが1987年に論文報告をしたのが、CRISPRが見出された最初の例です。この配列は、後の研究者によって、CRISPR、Clustered Regularly Interspaced Short Palindromic Repeatsと名付けられることになります。
Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. (1987). Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J Bacteriol 169: 5429-5433. PDFリンク
石野博士らは、大腸菌(E. coli)のiap遺伝子の構造を調べた際に、iap遺伝子の近傍に不思議な配列があることを見出しました。29塩基からなる共通配列が、32塩基からなるスペーサー配列を挟むようにして、5回繰り返し配置されていたのです。この共通配列の内部はパリンドーム(回文構造)になっていました。
FIG. 5. Comparison of direct-repeat sequences consisting of 61 base pairs in the 3′-end flanking region of iap. The 29 highly conserved nucleotides, which contain a dyad symmetry of 14 base pairs (underlined), are shown at the bottom. Homologous nucleotides found in at least two DNA segments are shown in boldface type. The second translational termination codon is boxed. The nucleotide numbers are in parentheses.
An unusual structure was found in the 3′-end flanking region of iap (Fig. 5). Five highly homologous sequences of 29 nucleotides were arranged as direct repeats with 32 nucleotides as spacing. The first sequence was included in the putative transcriptional termination site and had less homology than the others. Well-conserved nucleotide sequences containing a dyad symmetry, named REP sequences, have been found in E. coli and Salmonella typhimurium (28) and may act to stabilize mRNA (18). A dyad symmetry with 14 nucleotide pairs was also found in the middle of these sequences (underlining, Fig. 5), but no homology was found between these sequences and the REP sequence. So far, no sequence homologous to these has been found elsewhere in procaryotes, and the biological significance of these sequences is not known.
この論文では、「現在のところ、今回見つかった配列と相同性を示す配列は他の原核生物では見つかっていない。また、この配列の生物学的な意義は不明である(So far, no sequence homologous to these has been found elsewhere in procaryotes, and the biological significance of these sequences is not known)」と述べています。
石野博士らが大腸菌で初めて見つけたこの不思議な構造が、実は他の細菌(Bacteria)や古細菌(Archaea)にも存在していることが、他の研究者らによって徐々に明らかにされていき、Mojicaらが、DNA配列のこの特徴的な構造が実は原核生物のゲノムにおいては広く一般的に存在しているのだということを2000年に報告しました。
Mojica, F.J.M., Dıez-Villasenor, C., Soria, E., and Juez, G. (2000). Biological
significance of a family of regularly spaced repeats in the genomes of Archaea,
Bacteria and mitochondria. Mol. Microbiol. 36, 244–246. HTMLリンク
この論文で、Mojicaらはこの不思議な配列をShort Regularly Spaced Repeats (SRSRs)と名づけましたが、定着しませんでした。また、論文の結論は、多くの原核生物で見出されたのだから何か共通する機能があるのではないかという問題提起にとどまっています。
The question emerges here as to whether the SRSRs have a common function in prokaryotes, or whether their presence is reminiscent of ancient sequences and their role diverged with evolution. The universality, phylogeny and biological significance of this peculiar family of repeats arises as an item to be elucidated. (Mojica eta l., 2000 結論のセクションより)
Cas遺伝子の発見
CRISPRという繰り返し配列が原核生物のゲノム中に広く存在することはわかりましたが、その配列の意味するところはいっこうにわからないままでした。このミステリーを解き明かす手がかりを与えたのが、Jensenらによる、CRISPR近傍に存在する遺伝子群の発見です。
Identification of genes that are associated with DNA repeats in prokaryotes. Jansen R, Embden JD, Gaastra W, Schouls LM. Mol Microbiol. 2002 Mar;43(6):1565-75. PDF link
Cas遺伝子(Cas1, Cas2, Cas3, Cas4)を報告したこの論文では、奇妙な繰り返し配列をCRISPRと名付けており、この呼称が定着しています。
To appreciate their characteri-stic structure, we will refer to this family as the clustered regularly interspaced short palindromic repeats (CRISPR). In most species with two or more CRISPR loci, these loci were flanked on one side by a common leader sequence of 300-500 b. (中略)Four CRISPR-associated (cas) genes were identified in CRISPR-containing prokaryotes that were absent from CRISPR-negative prokaryotes. The cas genes were invariably located adjacent to a CRISPR locus, indicating that the cas genes and CRISPR loci have a functional relationship. The cas3 gene showed motifs characteristic for helicases of the superfamily 2, and the cas4 gene showed motifs of the RecB family of exonucleases, suggesting that these genes are involved in DNA metabolism or gene expression. (Jansen et al., Mol Microbiol. 2002 Mar;43(6):1565-75.の論文の要旨から一部抜粋)
Cas遺伝子がヘリカーゼやエクソヌクレアーゼといったDNAの代謝に関わる酵素のモチーフを持っていたことから、この論文では、この謎の繰り返し配列を作るのにこれらのCasタンパク質が関与しているのではないかと、正しく推測していました。
The finding in this study that the CRISPR loci were strictly associated with a set of homologous genes, one of which has nucleic acid helicase motifs (the Cas3 homologues), one of which has exonuclease activity (the Cas4 homologues) and one of which has a high pI (the CasI homologues), as is often found for DNA-binding proteins, may be suggestive of a role for the Cas proteins in the genesis of CRISPR loci. (Jansen et al., Mol Microbiol. 2002 Mar;43(6):1565-75.の論文の議論のセクションから抜粋)
CRISPRスペーサー配列の正体
繰り返し配列の間に挟みこまれた配列の起源があいかわらず謎でしたが、ほどなくして、これがファージの配列だということがわかってきました。その結果、CRISPRはバクテリアが外敵であるファージから身を守るための仕組みなのではないかという推測がなされました。
CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. C. Pourcel, G. Salvignol, G. Vergnaud. First Published Online: 01 March 2005, Microbiology 151: 653-663. HTML link
One possible explanation for that finding could be that CRISPRs are structures able to take up pieces of foreign DNA as part of a defence mechanism. In this view, it is tempting to further speculate that CRISPRs may represent a memory of past ‘genetic aggressions’. The fact that most of the spacers described in other bacteria have no homologue in the databases could still be explained by such a phage origin, as only a very small number of the existing bacteriophages have so far been sequenced. (Pourcel et al., Microbiology 151: 653-663. ディスカッションセクションより抜粋)
Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Bolotin A, Quinquis B, Sorokin A, Ehrlich SD. Microbiology. 2005 Aug;151(Pt 8):2551-61. HTML link
Here we report a correlation between the number of spacers in a locus and the resistance of S. thermophilus to phage infection, suggesting that CRISPRs can have a different biological role, protecting the bacteria against phage attack. (Bolotin et al., Microbiology 151(Pt 8):2551-61)
CRISPR-Casシステムの作用機序と生物学的な意味の解明
CRISPRの配列の中の、スペーサー配列の正体がファージなど外来ゲノム断片であることがわかり、CRISPR-Casシステムはいわばバクテリアにとっての免疫システムだと予想され、実際にそのように働くことが実証されました。
CRISPR provides acquired resistance against viruses in prokaryotes. Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. Science 2007 Mar 23;315(5819):1709-12.
Clustered regularly interspaced short palindromic repeats (CRISPR) are a distinctive feature of the genomes of most Bacteria and Archaea and are thought to be involved in resistance to bacteriophages. We found that, after viral challenge, bacteria integrated new spacers derived from phage genomic sequences. Removal or addition of particular spacers modified the phage-resistance phenotype of the cell. Thus, CRISPR, together with associated cas genes, provided resistance against phages, and resistance specificity is determined by spacer-phage sequence similarity. (Barrangou et al., 2007 要旨)
Cas9–crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria.
Here, we demonstrate that the Cas9–crRNA complex of the Streptococcus thermophilusCRISPR3/Cas system introduces in vitro a double-strand break at a specific site in DNA containing a sequence complementary to crRNA. DNA cleavage is executed by Cas9, which uses two distinct active sites, RuvC and HNH, to generate site-specific nicks on opposite DNA strands. Results demonstrate that the Cas9–crRNA complex functions as an RNA-guided endonuclease with RNA-directed target sequence recognition and protein-mediated DNA cleavage. (要旨より一部抜粋)
CRISPRの遺伝子編集技術への応用
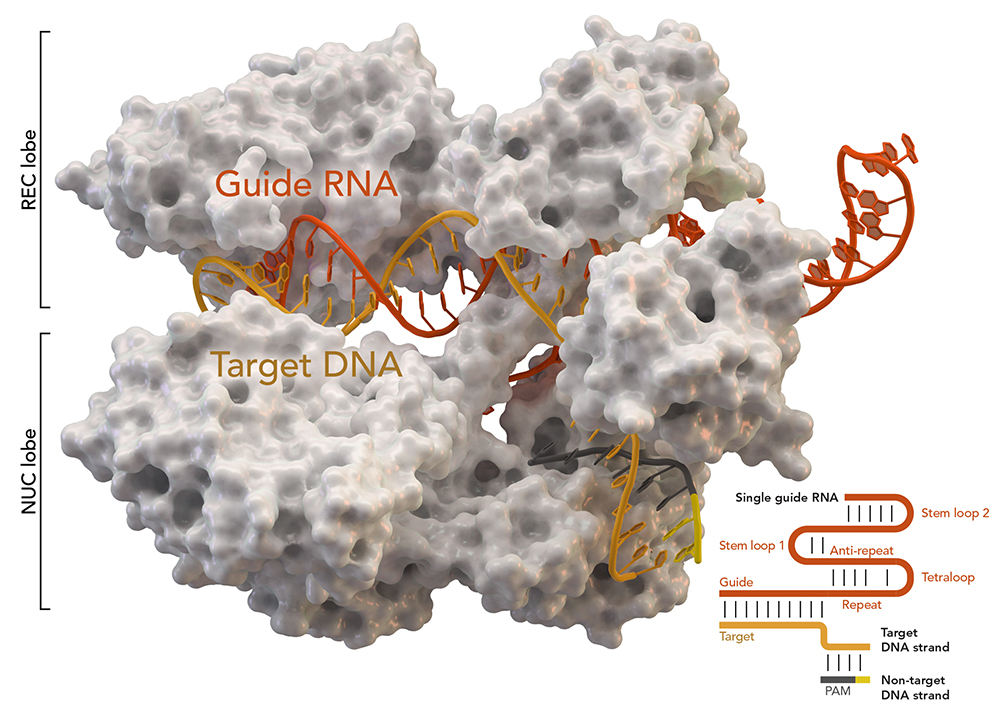
バクテリアなどの原核生物が持っているCRISPR/Casシステムにおいて、標的DNA配列を特異的に認識され、標的DNAが正確に切断されることがわかると、ゲノム編集ツールとしての有用性が直ちに認識されました。Casタンパク質のグループには、複合体を作って働くものや、ひとつで全ての役割を備えたものなど種類があります。Cas9は一人で全ての役割をこなす巨大なタンパク質で、これとガイドRNA(クリスパーRNA(標的配列)とトレーサーRNAを、人工的に一本鎖にまとめたもの)の2つのプレーヤーだけでゲノム編集ができてしまうという驚くべき簡便さが、応用の広さを生み出しました
A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity.
Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. Science. 2012 Aug 17;337(6096):816-21.
We propose an alternative methodology based on RNA-programmed Cas9 that could offer considerable potential for gene-targeting and genome-editing applications. (Jinek et al., 2012)
George Church博士やFeng Zhang博士の研究グループにより、CRISPR/Cas9システムが真核生物においても働くことが示され、ゲノム編集ツールとして人間を含めた全ての真核生物に応用する道が開かれました。
RNA-Guided Human Genome Engineering via Cas9. Prashant Mali, Luhan Yang, Kevin M. Esvelt, John Aach, Marc Guell, James E. DiCarlo, Julie E. Norville, George M. Church. Science 15 Feb 2013; Vol. 339, Issue 6121, pp. 823-826
The fully defined nature of this two-component system suggested that it might function in the cells of eukaryotic organisms such as yeast, plants, and even mammals. By cleaving genomic sequences targeted by RNA sequences (4–6), such a system could greatly enhance the ease of genome engineering. (Mali et al., 2013 のイントロダクションより。太字強調は当サイト)
Multiplex Genome Engineering Using CRISPR/Cas Systems. Le Cong, F. Ann Ran, David Cox, Shuailiang Lin, Robert Barretto, Naomi Habib, Patrick D. Hsu, Xuebing Wu, Wenyan Jiang, Luciano A. Marraffini, Feng Zhang. Science 15 Feb 2013:Vol. 339, Issue 6121, pp. 819-823
KS Community Lecture: Genome Editing Using CRISPR-Cas Systems
フェン・ジャン(Feng Zhang)博士による解説
- Development and Applications of CRISPR-Cas9 for Genome Engineering. Patrick D.Hsu, Eric S.Lander, Feng Zhang. Cell Volume 157, Issue 6, 5 June 2014, Pages 1262-1278 HTML link
- MIT Technology Day 2017: Professor Feng Zhang (レクチャー YOUTUBE動画18:14)
ちなみに、フェン・ジャン(Feng Zhang)博士は、大学院時代にスタンフォード大学のカール・ダイセロス博士のラボでオプトジェネティクスに関する仕事をしています。
Zhang studied chemistry and physics at Harvard and graduated with the highest honors. He then headed to Stanford University for his doctoral work, where he joined the newly formed lab of Karl Deisseroth, who had just begun to develop optogenetics as a method for manipulating brain activity. Over the next five years, Zhang played a central role in making optogenetics a reality.
In 2009, after earning a PhD in chemistry, Zhang switched his focus to genome editing. That same year, he received a prestigious three-year Harvard Junior Fellowship, during which he worked in the laboratories of two Harvard Medical School professors, Paola Arlotta and George Church. There, he helped develop a new method of gene editing through the adaptation of TAL effectors (TALEs), sequence-specific DNA-binding proteins found in plant pathogens that alter gene expression in plants.
Zhang joined the MIT faculty in January 2011 and became a core member of the Broad Institute of MIT and Harvard. (https://lemelson.mit.edu/winners/feng-zhang)
ダウドナ博士によるCRISPRに関する基本事項の解説
- Biology and Applications of CRISPR Systems: Harnessing Nature’s Toolbox for Genome Engineering. Addison V.Wright, James K.Nuñez, Jennifer A.Doudna. Cell Volume 164, Issues 1–2, 14 January 2016, Pages 29-44. PDF link
Jennifer Doudna: CRISPR Basics (2017 CRISPR Workshop YOUTUBE動画 Innovative Genomics Institute – IGI 2017/11/04)
CRISPRの発見者は誰?
CRISPRという自然現象の謎解きには多くの研究者が関わっています。また、CRISPR/Cas9を有効なゲノム編集技術のツールにするための技術開発も複数の研究グループが熾烈な競争を繰り広げています。そのため、誰がCRISPRの発見者なのか、誰が一番大きな貢献をしたといえるのかについては、見解が分かれるかもしれません。2017年度のJapan Prize(日本国際賞)は、「生命科学分野」ではエマニュエル・シャルパンティエ博士とジェニファー・ダウドナ博士の両氏が受賞しています。
シャルパンティエ博士とダウドナ博士は、「生命科学」分野において「CRISPR-Casによるゲノム編集機構の解明」という顕著な功績をあげたことが受賞理由となりました。両氏によって発表されたCRISPR-Casシステムによるゲノム編集は、遺伝子工学において従来方法と比べてはるかに安価で時間をとらず、圧倒的に容易な革命的な新技術です。どんな生物においても目的とするDNAを任意の部位で切断し、削除、置換、挿入など自在な編集を可能にしました。本技術は生命科学研究のツールとして爆発的に広がっただけでなく、育種、創薬、医療など幅広い分野で応用研究が進んでいます。(Japan Prize News No.57 Feb. 2017 PDFリンク)
また、2018年度カブリ賞ナノサイエンス分野では、エマニュエル・シャルパンティエ博士、ジェニファー・ダウドナ博士、および、リトアニアのヴィルギニユス・シクスニス (Virginijus Šikšnys)博士の3人が受賞しました。
2018 Kavli Prize Winners – NANOSCIENCE (World Science Festival YOUTUBE動画)
ゲノム編集技術CRISPR/Cas9の特許をめぐる熾烈な争い
特許に関しては、フェン・ジャン(Feng Zhang)博士ら(ブロード・インスティチュート)の陣営と、ジェニファー・ダウドナ博士(カリフォルニア大学バークレー校)らの陣営との間で熾烈な訴訟合戦が繰り広げられており、アメリカでの裁定とヨーロッパでの裁定が異なるなど、混沌とした印象があります。
The CRISPR Patent Battle: Implications for Downstream Innovation in Gene Editing
- 終わらないCRISPR特許論争、UCバークレーが不服申し立て(MIT Technology Review エミリー マリン (Emily Mullin) 2018.05.01, 16:58)
CRISPRの発見からツールとしての利用まで
- The Heroes of CRISPR (Eric S. Lander, Cell 164, January 14, 2016) 研究者に焦点を当てた、CRISPR発見の裏話的な内容
CRISPR関連の略語、用語
CRISPR: clustered regularly interspaced palindromic repeats (Jansen et al., 2002 Mol Microbiol)
spacer ( スペーサー ): CRISPRに保存されたウイルスDNAの小さな断片 (参考:PDBj)
Cas: CRISPR-associated protein (Jansen et al., 2002 Mol Microbiol)
PAM: protospacer adjacent motif
crRNA: CRISPR RNA (Brouns et al., 2008 Science) 標的となる外来遺伝子配列の断片であるスペーサー配列を含む。
tracrRNA: trans-acting CRISPR RNA (Deltcheva et al., 2011 Nature) Casタンパク質と結合するための足場としての役割を担う部分。
gRNA: guide RNA (sgRNA (single-strand guide RNA, single guide RNA, short guide RNA)とも表記) 自然界では複合体を形成して働くcrRNAとtracrRNAを、ゲノム編集ツールとして利用しやすいよう、人工的にひと続きのRNAにしたもの。
Cascade: CRISPR-Associated Complex for Antiviral Defense (Brouns et al., 2008 Science) タンパク質からなる巨大な複合体で内部にCRISPR配列のRNA転写物があり、これを使って感染によるウイルスDNAと適合するものがいないか監視している。(参考:PDBj)
Cas9: Cascade 9 (もしくはCRISPR associated protein 9)(参考: Roy et al., 2018 Front Genet.)2018 Cas9は、II型CRISPR系に分類される。監視タンパク質と切断を実行する部分の両方が1本のタンパク質鎖の中に収められている。(参考:PDBj) cas9 (formerly named “cas5” or “csn1”) is the signature gene for type II systems (8)(出典:Gasiunas et al., 2012;PNAS 109(39)E2579-E2586)
一般読者向けCRISPR解説記事
- CRISPR Part 1: A Brief History of CRISPR (TWIST BIOSCIENCE Dec 12, 2017)
遺伝子編集技術CRISPR-Casシステムの技術的な問題点
- ゲノム遺伝子の欠失には非常に効率の良い方法ではあるが、塩基置換や挿入の効率はまだ十分ではなく、目的の変異の導入には多くの受精卵または胚が必要である。
- 現在使用されているガイドRNAが認識するDNA配列は20塩基であるため、特異性はそれほど高くなく、目的としないゲノム配列にも変異が導入される可能性は否定できない。
(http://www8.cao.go.jp/cstp/tyousakai/life/haihu94/shiryo2-2.pdf)
遺伝子編集技術CRISPR-Casシステムの社会的な問題点
動物や植物の品種改良技術として使われる可能性が高い。その場合に、原理的には自然突然変異と区別がつかな
いが、遺伝子組換え食品として扱わないかは、合意が得られていない。(http://www8.cao.go.jp/cstp/tyousakai/life/haihu94/shiryo2-2.pdf)
CRIPR-Casの種類と分類
- Annotation and Classification of CRISPR-Cas Systems. Kira S. Makarova and Eugene V. Koonin. Methods Mol Biol. 2015; 1311: 47–75. PDF link
- Cpf1 Is a Single RNA-Guided Endonuclease of a Class 2 CRISPR-Cas System. BerndZetsche, Jonathan S.Gootenberg, Omar O.Abudayyeh, Ian M.Slaymaker, Kira S.Makarova, PatrickEssletzbichler, Sara E.Volz, JuliaJoung, Johnvan der Oost, AvivRegev, Eugene V.Koonin, Feng Zhang. Cell
Volume 163, Issue 3, 22 October 2015, Pages 759-771. Here, we report characterization of Cpf1, a putative class 2 CRISPR effector. We demonstrate that Cpf1 mediates robust DNA interference with features distinct from Cas9. Cpf1 is a single RNA-guided endonuclease lacking tracrRNA, and it utilizes a T-rich protospacer-adjacent motif. Moreover, Cpf1 cleaves DNA via a staggered DNA double-stranded break. - Expanding the catalog of cas genes with metagenomes. Quan Zhang, Thomas G. Doak, and Yuzhen Ye. Nucleic Acids Res. 2014 Feb; 42(4): 2448–2459.
- Evolution and classification of the CRISPR–Cas systems. Makarova et al., 2011. Nature Reviews Microbiology volume 9, pages 467–477(無料Abstract)
バイオ関連企業による研究者向けのCRISPR-Cas技術解説
- 1. An Intro to CRISPR Cas9 (abmgood.com 6:21動画)
レビュー論文
- CRISPR/Cascade 9-Mediated Genome Editing-Challenges and Opportunities. Bhaskar Roy, Jing Zhao, Chao Yang, Wen Luo, Teng Xiong, Yong Li, Xiaodong Fang, Guanjun Gao, Chabungbam O. Singh, Lise Madsen, Yong Zhou, and Karsten Kristiansen. Front Genet. 2018; 9: 240.
- Genome editing: A breakthrough in life science and medicine. Izuho Hatada and Takuro Horii. Endocrine Journal 63(2):105-110. (2016) PDFリンク
- 総説 ゲノムから見た最近の進化ーCRISPによる生存戦略ー 中川一路 Dental Medicine Research 33(3):236-241. (2013). PDFリンク
原著論文
数が多いため主要な論文のみ挙げています。
CRISPRの発見
- Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. (1987). J Bacteriol 169: 5429-5433. PDFリンク
- Unusual Nucleotide Arrangement with Repeated Sequences in the Escherichia coli K-12 Chromosome. ATSUO NAKATA,* MITSUKO AMEMURA, AND KOZO MAKINO. Department of Experimental Chemotherapy, Research Institute for Microbial Diseases, Osaka University, 3-1, Yamadaoka, Suita, Osaka, Japan 565. Received 19 December 1988/Accepted 13 March 1989 JOURNAL OF BACTERIOLOGY, June 1989, Vol. 171, No. 6 p. 3553-3556 PDF link
- Long stretches of short tandem repeats are present in the largest replicons of the Archaea Haloferax mediterranei and Haloferax volcanii and could be involved in replicon partitioning. F.J.M. Mojica C. Ferrer G. Juez F. Rodríguez‐Valera. molecular microbiology July 1995;17(1):85-93
- Biological significance of a family of regularly spaced repeats in the genomes of Archaea,
Bacteria and mitochondria. Mojica, F.J.M., Dıez-Villasenor, C., Soria, E., and Juez, G. (2000). Mol. Microbiol. 36, 244–246. PDFリンク
Casの発見
- Identification of genes that are associated with DNA repeats in prokaryotes. Jansen R, Embden JD, Gaastra W, Schouls LM. Mol Microbiol. 2002 Mar;43(6):1565-75. PDF link
CRISPRが働くメカニズム と生物学的な意義(細菌に備わる防御機構)
- CRISPR provides acquired resistance against viruses in prokaryotes. Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. Science 2007 Mar 23;315(5819):1709-12.
- Cas9–crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria.
ゲノム編集技術としてのCRISPR
- A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. Science 2012 Aug 17;337(6096):816-21.
- Multiplex Genome Engineering Using CRISPR/Cas Systems. Le Cong, F. Ann Ran, David Cox, Shuailiang Lin, Robert Barretto, Naomi Habib, Patrick D. Hsu, Xuebing Wu, Wenyan Jiang, Luciano A. Marraffini, Feng Zhang. Science 15 Feb 2013; Vol. 339, Issue 6121, pp. 819-823
CRISPRに関する最近の論文
- C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector.
Omar O. Abudayyeh, Jonathan S. Gootenberg, Silvana Konermann, Julia Joung, Ian M. Slaymaker, David B.T. Cox, Sergey Shmakov, Kira S. Makarova, Ekaterina Semenova, Leonid Minakhin, Konstantin Severinov, Aviv Regev, Eric S. Lander, Eugene V. Koonin, Feng Zhang. Science 02 Jun 2016. - Direct CRISPR spacer acquisition from RNA by a natural reverse transcriptase–Cas1 fusion protein. Sukrit Silas, Georg Mohr, David J. Sidote, Laura M. Markham, Antonio Sanchez-Amat, Devaki Bhaya, Alan M. Lambowitz, Andrew Z. Fire. Science 26 Feb 2016;Vol. 351, Issue 6276
その他のCRISPR-CASに関する論文
- Protospacer adjacent motif (PAM)-distal sequences engage CRISPR Cas9 DNA target cleavage.
Cencic R, Miura H, Malina A, Robert F, Ethier S, Schmeing TM, Dostie J, Pelletier J. PLoS One. 2014 Oct 2;9(10):e109213.
参考
- Genome damage from CRISPR/Cas9 gene editing higher than thought July 16, 2018, phys.org
- CRISPR (ウィキペディア)
- Virginijus Šikšnys (Wikipedia)
- „Imam genų žirkles, iškerpam klaidą, ligos nelieka“ – Virginijus Šikšnys | Laikykitės ten pokalbiai(YOUTUBE動画トークショー)
- Lithuanian Lesson 2 – Alphabet (YOUTUBE) リトアニア語の発音
- Lesson One: Lithuanian Video Course (YOUTUBE) リトアニア語の発音