ImageJは無料の画像処理ソフトです。マックでもウインドウズでもリナックスのOSでも使えて、研究で必要な画像処理が比較的簡単にできるので、バイオ研究者の間で非常によく使われています。自分も長年にわたり使ってきましたが、画像データを見直したり簡単な解析(特定の領域の平均輝度を取得するなど)を行うときには、Image Jを使うのが一番手軽で便利な方法だと思います。
Fijiの勧め
実際には、Image Jよりもさらに高機能なFijiを自分は使っています。Fijiは単にImageJそのもので(Fiji is just ImageJ)、プラグインが最初からたくさんくっついてくるだけです。Image Jは様々なデータ処理・データ解析のためにプラグインを入れることができますが、Fijiには最初からこれらのプラグインがてんこ盛りで入っていますので、いちいち後からインストールする手間がなくて良いです。顕微鏡メーカーやカメラメーカー独特の画像フォーマットでも、大抵の画像ファイルがFijiで開けてしまいます(Bio-Formatsを利用するなどして)。
Image Jのダウンロード
ImageJは、https://imagej.nih.gov/ij/download.html からダウンロードできます。Fijiは、https://imagej.net/Fiji/Downloads からダウンロードできます。
Image Jのインストール方法
インストール方法は、ダウンロード後の指示に従って行ってください。
Image Jの使い方
アイデア次第でいろいろな画像処理がImageJでできますが、自分がよくやる画像処理をいくつか紹介します。
Image Jを用いて画像を二値化する方法
元画像としてカラヤンの画像をウィキペディアから拝借。

メニューからImage, Adjust, Threshold…を選びます。2つのスライダーを調節して、望む画像になるような閾値を探して決めましょう。今の場合、76-255としました。Applyボタンをクリックして適用します。

ImageJを使って細胞の数を数える方法
サンプルとしてこの血球の画像をお借りします。
この画像を使って、ImageJでカラー画像を白黒にし、2値化して、粒子解析の機能を利用して、細胞の数を数えてみます。
メニューからFile, Open…として、ダウンロードしておいた目的の画像ファイルを開きます。
メニューからImage, Type, 8-bitと順に選びます。これで白黒画像に変更されました。

メニューからImage, Adjust, Threshold…を選びます。2つのスライダーを調節して、全ての細胞が分離できそうな閾値を探して決めましょう。今の場合、183-255としました。Applyボタンをクリックして適用します。
 (黒い部分の画素値が0で白い部分が255)
(黒い部分の画素値が0で白い部分が255)
さてそれでは粒子解析 (Particle Analysis) の機能を使って、数を数えましょう。
抽出された粒子(今の場合、赤血球)のパラメータとして何を知りたいかは、メニューからAnalyze, Set Measurements… とすることにより選択できます。パネルの中のAreaにチェックが入った状態にします。他は今はとりあえず不要です。ここで、写真の中の赤血球のだいたいの面積を知っておきましょう。メニュー上にあるROIの形(四角形や丸や不定形など)から丸(Oval)を選び、赤血球の一つがちょうど同じ大きさになるように合わせます。そして、Analyze, Measureと選ぶと、Resultsという名のウインドウ上に(みあたらなければ、メニューからWindow,Resultsと選ぶと、最前面に表示されます)Areaが表示されます。今の場合420という数値になりました。カーソルを細胞の上にあてると画素の値が出ますが、今やってみると、黒く表示されている赤血球の画素の値が0で、白いbackグランウンドの値が255でした。粒子を数えるときは値が255でないと困るので、ここでメニューからEdit, Invertを選び白黒を反転させます。
 (白い部分の画素が255、黒が0)
(白い部分の画素が255、黒が0)
それではいよいよ粒子解析を行うため、Analyze, Analyze Particles…を選びます。粒子としてカウントするサイズをここで選ぶことができます。ここで粒子の面積のサイズが取りそうな範囲を適切に設定してやれば、画面上のノイズや目的外のものを除外できる可能性があります。さきほど赤血球の面積がおよそ400画素程度だったので、ここでは広めにとって、Size (pixel^2): 200-800 としておきましょうか。Circularityは全範囲にしておきます。明らかに丸っこいものしかないというのであれば、ここで強い条件(1に近い範囲)を設定してやれば、不定形のごみを区別して除くこともできます。あとは、Display results, Clear results, Summarize, Include holesにチェックを入れておきます。 Include holesというのは、赤血球の丸い形の真ん中が2値画像にしたときに抜けていても、その穴は埋めた状態として(つまり穴に見える部分の画素も細胞に属する画素として)考えてくれるということです。あと、Show:Masksを選んでおきます。粒子解析を実行した結果、マスク画像として以下が得られました。ここには、粒子としてカウントされたものが表示されています。たしかに「穴」も埋められていることがわかります。

さて、Summaryというウインドウに結果が表示されており、それを見るとCountが573、Average Size(粒子の面積の平均)が419.211となりました。つまり、赤血球の数がおよそ573個あって、それらの面積の平均は419画素だったというわけです。ちなみに、もとのカラー写真をみると白血球みたいなものも一つありましたが、この画像処理ではそれは無視されて赤血球と区別がつかなくなっているため、カウントされています。
ImageJを使ってくっついている細胞同士を分離する方法
こんなふうに細胞同士がくっついて見える写真で数を数えるにはどうすればいいのでしょうか?
 (元画像のリンク)
(元画像のリンク)
メニューの欄からImage, Type, 8-bitと選び、まず白黒画像にします。
 8-bit
8-bit
Image, Adjust, Threshold…で2値化します。スライダーをうごかして、できるだけ細胞がはっきり見えるように調整しました。細胞の画素が0で背景が255になっていたので、Edit, Invertで白黒を反転させます。
 Threshold
Threshold
Process,Binary, Fill Holesで細胞の真ん中部分の「穴」を埋めます。まあ完全ではありませんが。これくらいなら、手作業で不完全に残った「穴」の開いた細胞を埋めてもいいかもしれません。
 Fill Holes
Fill Holes
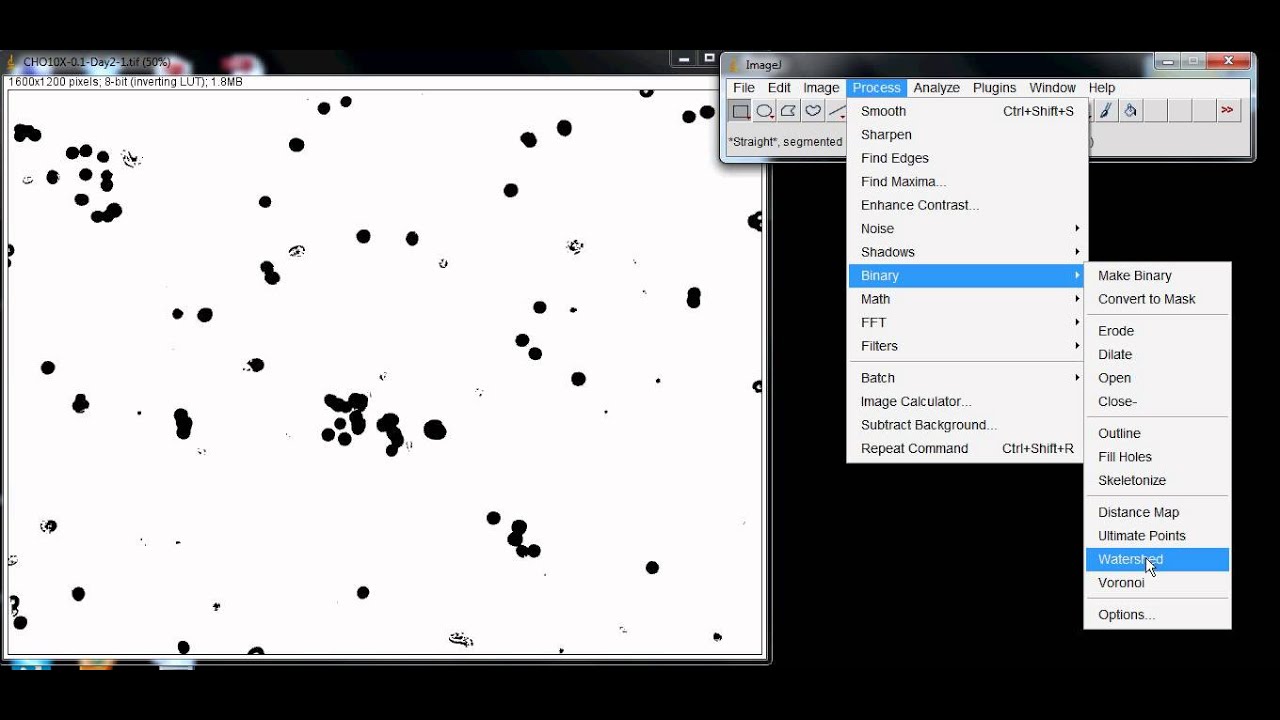
さて、この画像は結構細胞同士がくっついていますが、Process, Binary, Watershed により、無理やり分離させてみます。
 Watershed
Watershed
完全とはいいがたいですが、大半に関してくっついていた細胞が分離できています。これの画像に対して視野を覆うように円形のROIを適用してParticle Analysisを適用すれば、細胞数が数えられるでしょう。
- Basic Intensity Quantification with ImageJ (PDF) If you have particles that have merged together, Process →Binary→Watershed can often (but not always) accurately cut them apart by adding a 1 pixel thick line where it feels the division should be. *2つの粒子がくっついている場合にウォーターシェッドというアルゴリズムを用いて分離する機能もImageJにはあるようです。
Watershedの機能も用いて細胞を分離して、細胞数をカウントする方法を示したYOUTUBE動画があったので紹介しておきます。
Counting Cells with ImageJ by Kevin Foley 2013/09/10
Image Jを用いて蛍光標識された多数の物体の面積を求める方法
たまたま検索でヒットした論文の図をお借りして、調べたい構造の面積をImageJを使って求めてみます。Fig.1Dに蛍光画像があるのでそれを出発材料にします。
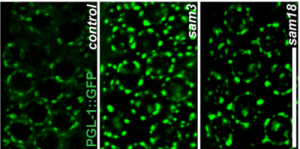
まずは白黒にしましょう。ImageのメニューでImage, Type, 8-bitと選びます。
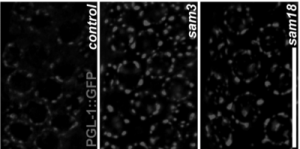
さて、蛍光標識された細胞内の構造物の大きさ(画像上の面積)の絶対的な数値を求めるのは困難でしょう。閾値の設定しだいでいくらでも面積が変わってきてしまいます(標識された部分と標識されていない部分の輝度に十分の差があればそこで区切れますが)。この3枚の写真が同じ撮影条件だったとして、一つの閾値を3枚に適用することにして、一番もっともらしいと主観で判断した閾値にしてみます。Image, Adjust, Threshold… としてスライダーで適当な値を選びました。
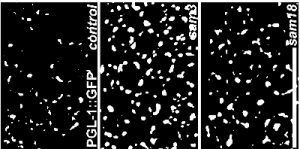
これでおのおのの「粒子」の面積を計算してみます。写真にラベルがあって邪魔なのでその部分は除いて計算します。そのためには解析したい範囲だけ四角いROIを設定します。Analyze, Set Measurements…でまず計算したいパラメータとしてAreaにチェックを入れて他は今は不要なので外しておきます。そして、Analyze, Analyze Particles…を選び、Size pixel^2):1-Infinity, Cirularity:0.00-1.00, Show:Masksとして、Display results, Clear results, Summarize, Include holesにはチェックをいれておきます。これでOKをクリックすれば、計算が行われます。ROIを次の写真に移動させて同様に計算していきましょう。
Controlの写真の解析結果は、Count:154, Average Size:92.526となりました。Results(個々の粒子の面積のデータ)をResultsウインドウ上のメニューからFile, Save As…で保存しておきます。
Sam3の写真の解析結果は、Count:287, Average Size:165.491となりました。同様に、結果を保存します。
Sam18の写真の解析結果は、Count:202, Average Size:144.337となりました。これも結果を保存します。
この論文で具体的に蛍光写真からどうやって面積を計算したのかは読んでいないのでわかりませんが、まあだいたいの傾向は論文のグラフと合っているのでよしとしましょう。
-
ELLI-1, a novel germline protein, modulates RNAi activity and P-granule accumulation in Caenorhabditis elegans PLOS Genetics
- P granules Jennifer T. Wang and Geraldine Seydoux Curr Biol. 2014 Jul 21; 24(14): R637–R638. Author manuscript
Image Jを用いて輝度を調べる方法
実験群と対照群に関して、何かの遺伝子発現の変化を、GFPリポーターを発現する細胞の蛍光写真で見比べて定量しようという実験がありがちだと思います。このような定量に関しては、蛍光写真撮影時に蛍光強度が飽和しないようなカメラの露光条件だったか(サンプルの蛍光強度と画像上の画素数が本当に比例するのか?ある明るさ以上は飽和してしまっていないか)、実験群と対照群の撮影条件は同一だったのか等のCRITICALなポイントが前提として存在します。そういったことがいい加減だと、ImageJで定量したつもりになっても、正しいデータ処理をしたことにはなりません。結果的に間違った結論を出す恐れがありますので注意が必要です。
- A Simple Method for Measuring Intracellular Fluorescence By Anthony Federico. Fortunately for microscopy users, measuring intracellular fluorescence has been made relatively simple through an ImageJ plugin called the Cell Magic Wand.
- Measuring cell fluorescence using ImageJ. Contributed by Martin Fitzpatrick, University of Birmingham, United Kingdom スナップショット付きの説明なのでわかりやすい。
- Analyzing fluorescence microscopy images with ImageJ. Peter Bankhead. Queen’s University Belfast. May 2014 (195 page PDF)
Image Jで画像にスケールバーを入れる方法
自分はスケールの写真を、サンプルを撮影したのと同じ条件(倍率)で撮影しておき、あとから画像の中の目盛りを長さを測るツールではかって算出しています。そしてその長さの長方形を画像に張り付けています(ROIをつくってカットするとバックグラウンドが白なら白く抜けるのでそれがスケールバーになる)。しかし、ピクセルと長さ(ミクロンなど)の関係をもとにスケールバーをつける機能がImageJに用意されています。
Analyze, Set Scale…を選びます。例えば20ミクロンが100ピクセルだとわかっていれば、Distance in pixels:100, Known distance: 20, Pixel aspect ratio:1, Unit of length: micronとします。すると、自動的に計算された結果として今の場合、Scale: 5 pixels/micronと表示されますので、OKをクリックします。
次に、画像を開いた状態で、Analyze, Tools, Scale bar…を選びますと、パネルが出現しますので表示させたいスケールバーの長さを例えばWidth in micron: 20, Height in pixels: 5, Color: White, Location:Lower Right などと選びOKをクリックします。すると画面右下に20ミクロンのスケールバーが表示されます。micronsとしていた単位は、写真に埋められたスケールバーにはギリシャ文字でμmと表示されました。

プレゼンテーションのスライド用なら数字と単位があったほうが便利でしょうから、これは便利な機能だと思います。Overlayがチェックされていなければスケールバーの長さを変更するとリアルタイムで反映されます。これでよしとなったら、Overlayにチェックを入れればよいでしょう。数字や単位を表示しないオプションは見当たりませんでした。
Image Jでマクロを使うことの勧め
研究においては大量の画像に対して同じ処理を適用するという作業が頻発することがあります。そのとき、ひとつひとつの画像に関して全て手作業で行うのはとても骨が折れますが、マクロを書いて一気に処理してしまうと楽ちんです。マクロを書くと言っても、ImageJにはマクロレコーダーという便利な機能がありますので、ゼロから自分で書く必要など全くなくて、マクロレコーダーをオンにした状態で自分がやりたい処理を一通りやってみて、マクロレコーダーが吐き出してくれるマクロをちょっと修正すれば良いのです。メニューから、Plugins, Macros, Record…と選べば、マクロレコーダーが起動します。プログラミングに苦手意識があって、マクロを使ったことがない人は若干敷居が高いと感じるかもしれませんが、一度使ってみればその便利さに驚き、手作業でやることがバカバカしくなること請け合いです。
Image Jの使い方に関する書籍(アマゾン)
- ImageJではじめる生物画像解析 三浦 耕太, 塚田 祐基 著 2016/3/26 学研プラス
- バイオ画像解析 手とり足とりガイド〜バイオイメージングデータを定量して生命の形態や動態を理解する! 小林 徹也, 青木 一洋 編集 2014/11/29 羊土社
『機械学習を生命科学に使う! シークエンスや画像データをどう解析し、新たな生物学的発見につなげるか? 』(実験医学増刊 Vol.38 No.20 小林徹也,杉村 薫,舟橋 啓/編 2020年12月14日発行) の第1章 機械学習入門 2.ImageJを使った機械学習による生物画像解析入門【三浦耕太】 にもImageJと機械学習を使った解析方法が紹介されていました。ImageJと機械学習、深層学習(ディープラーニング)とが組み合わせられるというのは知りませんでした。
- 機械学習を生命科学に使う!(羊土社)
この本を読むと、科学研究の手法の開発・普及の速さに驚かされ、また、自分が時代から取り残されたような気分がしました。研究者は、テクノロジーの進歩にキャッチアップするのが大変です。しかしそれを怠ればサバイブできません。
Image Jの使い方に関する参考サイト
- Bio-formats https://imagej.net/Bio-Formats
- imageJで画像にスケールバーを入れる 2013/8/20 LifeScienceProject
- スケールを設定しよう http://tec.miyakyo-u.ac.jp/hoshi/imagej2014/scale.htm
- 研究室での画像処理:ImageJの使い方・基礎編 2017.5.13 大金賢司 (26 page PDF)